引入
接上一篇的【有参估计】,这篇介绍无参估计,也就是说在这里我们事先不知道数据的模型,而要求数据进行划分,这也是实际中比较常见的情况。
这主要介绍三种无参估计方法,分别是:
1. 直方图(Histograms)
2. 核密度估计(Kernel Density Estimation,KDE)
3. K最邻近法(K-nearest Neighbors,KNN)
直方图(Histograms)
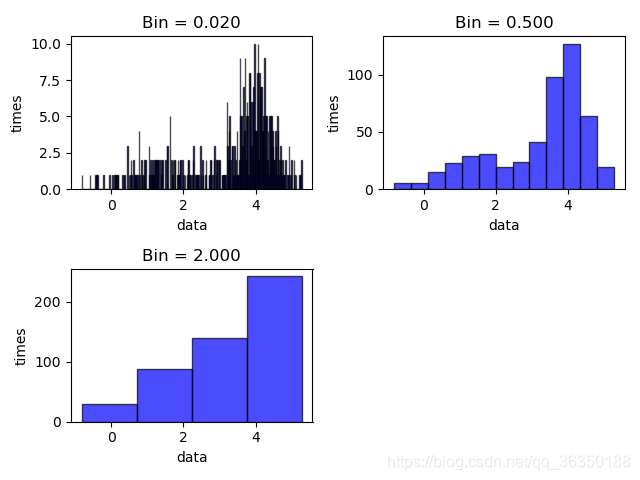
直方图是最简单的一种方法,通过对数据进行统计分类,画出直方图即可,这里需要注意的是直方图中$bin$值的选取。\
对于$bin$值的不同选取可能会有下面三种情况: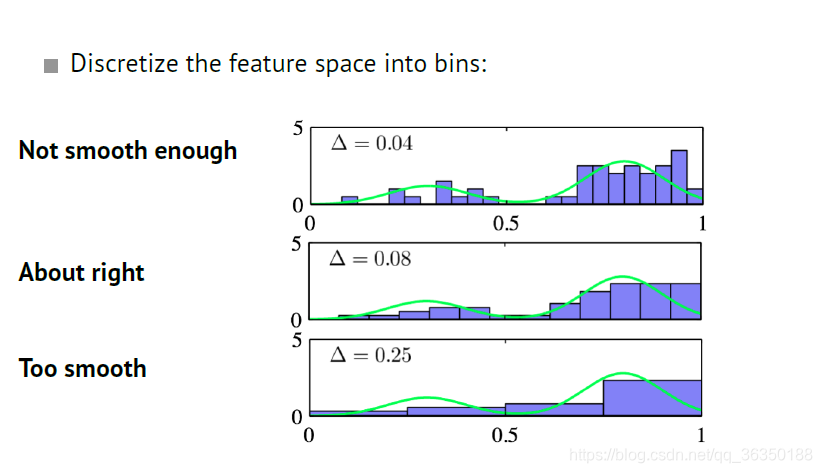
直方图的特点是:
- 使用非常普遍,没有数据的限制,任何概率密度都可以被任意地近似;\
- 这个方法太粗暴。。。
直方图的缺陷有:
- 高维空间中,$bin$的数目以指数级增长;\
- $bin$值得选取不容易。
直方图的相关计算:
(也不局限于直方图,这些计算方法是很通用的,后面也会用到。)\
我们现在有数据点$x$,其是从概率密度$p(x)$中取样的。\
$x$落在区域$R$的概率是:如果$R$非常小,体积为$V$,则$p(x)$几乎是常数:如果$R$很大,则:其中$N$是数据总数,$K$是落在区域$R$内的数据数。
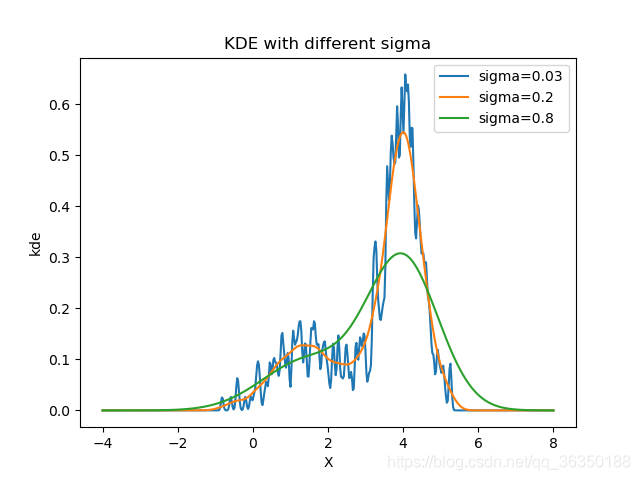
对于Kernel density estimation (KDE),是固定$V$并确定$K$,比如说:确定固定在超立方体中的数据点$K$的数量,如下图示:
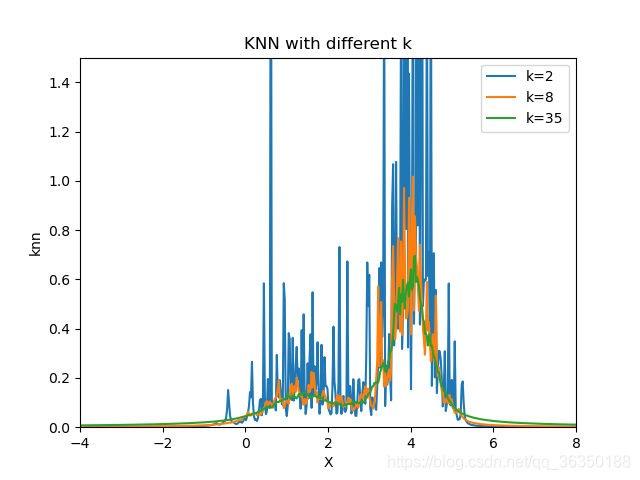
对于K-nearest neighbors (kNN),是固定$K$并确定$V$,比如说:增加球的尺寸直到$K$个点都落在球内,如下图:
KDE
Parzen Window
对于一个$d$维,边长为$h$的超立方体,我们有如下关系: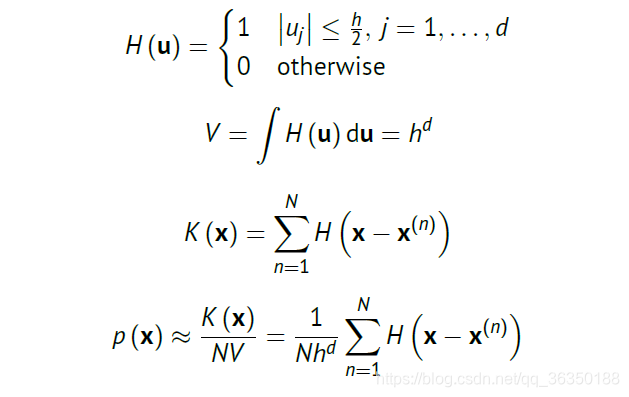
Gaussian Kernel
对于高斯核,我们则可以写成: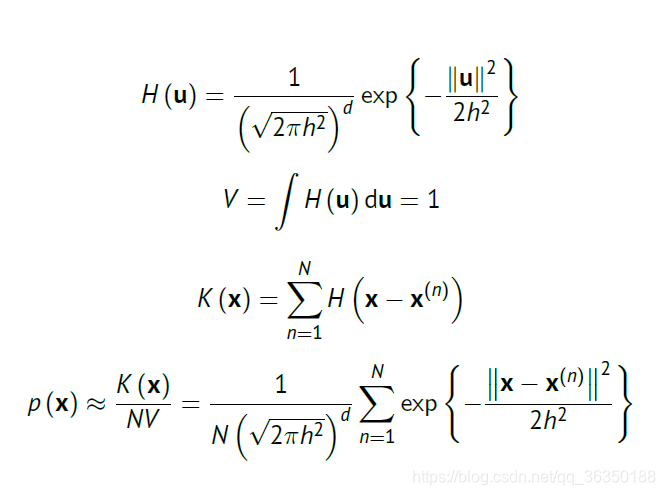
General Formulation – Arbitrary Kernel
对于更一般的形式,则有: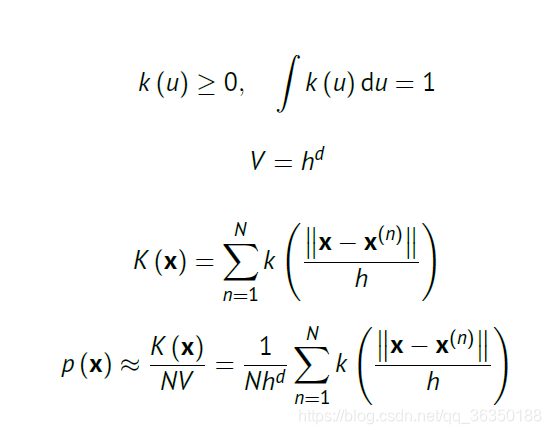
各种内核的总结
高斯核(Gaussian Kernel)
缺点是:
1. kernel has infinite support
2. Requires a lot of computation
Parzen window

缺点是:
1. Not very smooth results
Epanechnikov kernel

缺点是:
1. Smoother, but finite support
总结
核方法的缺点是:我们必须合适地选择核的带宽$h$。\
如果$h$太大则会过于平滑,太小则不够平滑。\
一个高斯核的例子: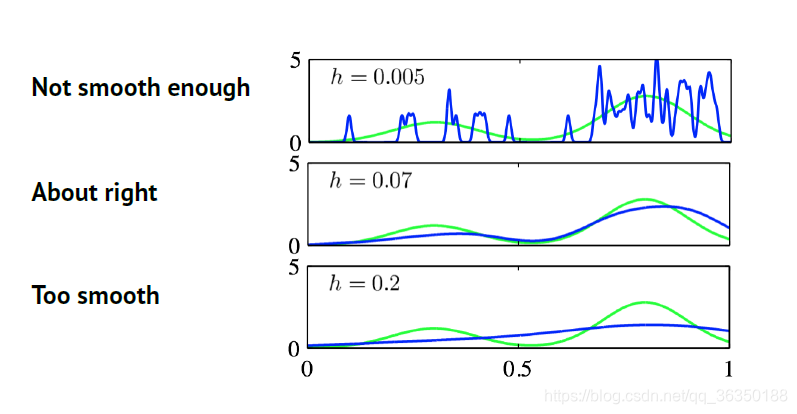
KNN
KNN分类大体意思是:假设我们的数据集有$N$个点,有$N_j$个点属于类$C_j$,并且有$\sum_jN_j=N$。现在要对点$x$进行分类,我们先画一个球,中心在$x$处,并且包含了(任意类的)$K$个点。假设球体积为$V$,且包含有$K_j$个点是属于$C_j$的,则我们有如下关系:
注意:与灰度图的$bins$和KDE的$h$类似,$K$值太大则会过于平滑,太小则不够平滑。
一个例子: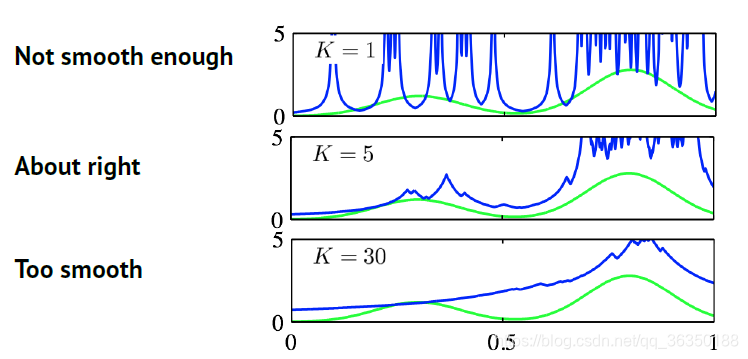
(附)作业相关代码

简单解释下作业:\
有两个数据集,分别为训练集和测试集,第1题要求画出不同$bins$下的直方图;第2题则是用高斯核,用不同的$\sigma$计算训练集的对数似然,比较差异性;第3题要求用不同的$K$值写$KNN$代码。\
代码如下:1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81
82
83
84
85
86
87
88
89
90
91
92
93
94
95
96
97
98
99
100
101
102
103
104
105
106
107
108
109
110
111
112
113
114
115
116
117
118
119
120
121
122
123
124
125
126
127
128
129
130
131
132
133
134
135
136
137
138
139
140
141import matplotlib.pyplot as plt
import numpy as np
from scipy.special import logsumexp
def load_data(path):
data = np.loadtxt(path)
return data
def plot_histogram(bins, arr):
if len(bins) % 2 == 0:
fir = len(bins) / 2 * 100
else:
fir = (len(bins) // 2 + 1) * 100
pos = fir + 20 + 1
interval = np.max(arr) - np.min(arr)
for i in range(len(bins)):
sub_bin = int(interval // bins[i]) + 1
plt.subplot(pos+i)
plt.hist(arr, bins = sub_bin, facecolor="blue", edgecolor="black", alpha=0.7)
plt.title("Bin = %.3f" % bins[i])
plt.xlabel('data')
plt.ylabel('times')
def kernel_density_estimate(x_arr, x_lst, sigma):
def _H_Gauss(h, u, d):
H_u = np.exp(-(np.linalg.norm(u) ** 2) / (2 * h * h)) \
/ (np.sqrt(2 * np.pi * h * h) ** d)
return H_u
def _K(N, x, x_arr, h, d):
k = 0
for i in range(N):
u = x - x_arr[i]
k += _H_Gauss(h, u, d)
return k
def p(N, x, x_arr, h, d, V):
p_x = _K(N, x, x_arr, h, d) / (N * V)
return p_x
V = 1
N = len(x_arr.flatten())
p_x_lst = []
d = len(x_arr.flatten())/(x_arr.shape[0])
for x in x_lst:
temp_p = p(N, x, x_arr, sigma, d, V)
p_x_lst.append(temp_p)
return p_x_lst
def _logsumexp(x):
return np.log(np.sum(np.exp(x)))
def ked_log_likelihood(x_arr, x_lst, sigma):
N = x_arr.shape[0]
ll_lst = []
for x in x_lst:
single_val = _logsumexp(-np.linalg.norm(x_arr.reshape(-1,1) - x, axis=1) ** 2 / (2 * sigma * sigma) \
- np.log(np.sqrt(2 * np.pi * sigma * sigma) * N))
ll_lst.append(single_val)
return np.sum(ll_lst)
def kNN(data, x_lst, k):
knn_lst = []
N = len(data.flatten())
for x in x_lst:
u = np.linalg.norm(x - data.reshape(-1,1), axis = 1)
r = np.sort(u)[k-1]
v = 2 * r
temp_p = k / (N * v)
knn_lst.append(temp_p)
return knn_lst, np.sum(np.log(knn_lst))
def Comparison_NPM(data, test_data, k_lst, sigma_lst):
for s in sigma_lst:
temp_kde_test = ked_log_likelihood(data, test_data, s)
temp_kde_train = ked_log_likelihood(data, data, s)
print('train_data: log_likelihood of KDE with sigma = %.2f: %.12f' % (s, temp_kde_train) )
print('test_data : log_likelihood of KDE with sigma = %.2f: %.12f' % (s, temp_kde_test) )
for k in k_lst:
_, temp_knn_test = kNN(data, test_data, k)
_, temp_knn_train = kNN(data, data, k)
print('train_data: log_likelihood of KDE with k = %d: %.12f' % (k, temp_knn_train) )
print('test_data : log_likelihood of KDE with k = %d: %.12f' % (k, temp_knn_test) )
def main():
path = "./dataSets/nonParamTrain.txt"
path_test = './dataSets/nonParamTest.txt'
data = load_data(path)
data_test = load_data(path_test)
bins_w = [0.02, 0.5, 2.0]
plot_histogram(bins_w, data)
plt.tight_layout()
plt.show()
num = len(data.flatten())
p_x = []
p_test = []
sigma = [0.03, 0.2, 0.8]
x_lst = np.linspace(-4, 8, data.shape[0])
for h in sigma:
temp_p = kernel_density_estimate(data, x_lst, h)
p_x.append(temp_p)
for x in p_x:
plt.plot(x_lst, x)
plt.xlabel("X")
plt.ylabel("kde")
plt.legend(['sigma=0.03','sigma=0.2','sigma=0.8'])
plt.title('KDE with different sigma')
plt.show()
k = [2, 8, 35]
knn_lst = []
for sub_k in k:
temp_knn, _ = kNN(data, x_lst, sub_k)
knn_lst.append(temp_knn)
for knn in knn_lst:
plt.plot(x_lst, knn)
plt.xlabel("X")
plt.ylabel("knn")
plt.axis([-4, 8, 0, 1.5])
plt.legend(['k=2','k=8','k=35'])
plt.title('KNN with different k')
plt.show()
Comparison_NPM(data, data_test, k, sigma)
if __name__ == "__main__":
main()
结果显示如下: