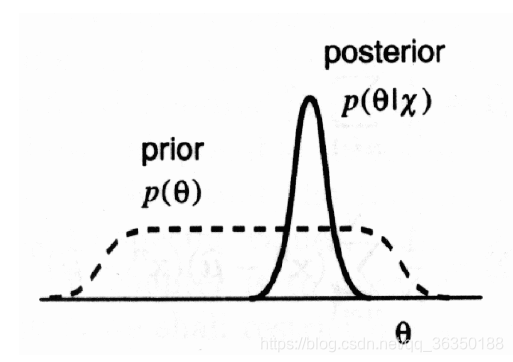
概率密度的引入
当我们有如下的点分布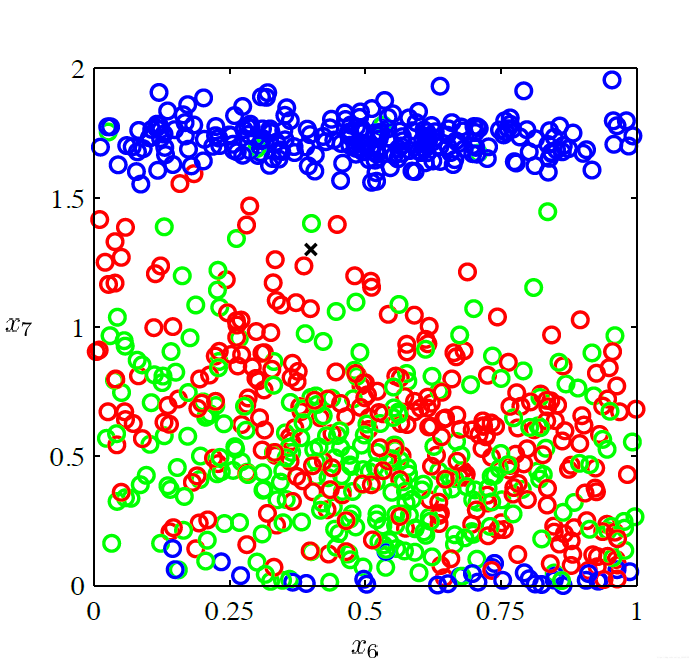
为了能区分它们,我们需要知道这些点的概率分布。常见的有贝叶斯最优分类(Bayes optimal classification),这是基于如下的概率分布:
其中的先验$p(C_k)$很容易算,就是统计,或者说数数,现在的问题就成了计算如下的条件概率密度:$p(x|C_k)$。这主要有三种情况:
1. Parametric model
2. Non-parametric model
3. Mixture models
(简单解释一下,有参估计是指我们知道样本数据符合某种概率密度模型,通过给出的数据求出所需要的参数,比如高斯分布的均值和方差,有参估计的Robust比较好;无参估计是指我们不知道这些数据点符合哪些模型,所以无法求出其参数,这种情况在现实生活中更加常见)
参数模型(Parametric model)
概率密度模型的符号表示为:
以高斯模型为例:
我们只要知道它的均值和方差就能完整地描述这个模型,即是:现在我们需要根据模型和数据集求出$\theta$。\
其似然函数表示为:($X$为数据集数据)
对于有参模型,这里介绍最大似然方法(Maximum Likelihood Method)
最大似然方法(Maximum Likelihood Method)
现假设我们有数据集$X=\left\{x_1,x_2,…,x_N\right\}$\
对于这些数据集我们假设数据是i.i.d(independent and identically
distributed)的,即是:
- $P(x_1\le\alpha, x_2\le\beta)=P(x_1\le\alpha)P(x_2\le\beta)$\
- $P(x_1\le\alpha)=P(x_2\le\alpha)$
通过该假设,似然计算可写为:
两边取对数为:
然后就是求导之类的事了。\
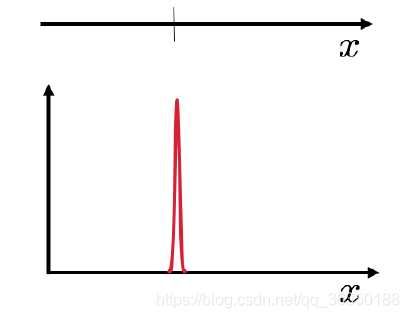
现在有个问题,如果$N=1$,即是说$X=\left\{x_1\right\}$,这时候产生的高斯模型会如下图所示:
看上去就像个$\delta$函数。\
这时候我们可以给平均数加上先验
贝叶斯估计(Bayesian Estimation)
与极大似然估计不同,贝叶斯估计假设待估计参数不是固定而是随机的。\
现在我们要根据给出的数据集$X$,得到$x$的密度函数$p(x)$。将其写成条件概率形式:用边缘概率和贝叶斯公式得出下图关系:
由于$p(x)$能根据$\theta$完全确定,即是说$\theta$是sufficient statistic,所以进而上面两个式子可写成:
(简单解释一下这个式子,$p(\theta |X)$表示估计参数对数据集$X$的依赖程度,也就是说$\theta$在一定程度上可以代指$X$,比如当$p(\theta|X)$在大多数地方都很小,但唯独在$\hat{\theta}$处很大,我们就能近似表示:$p(x|X) \approx p(x|\hat{\theta})$,该点也称为Bayes point)\
继续计算如下: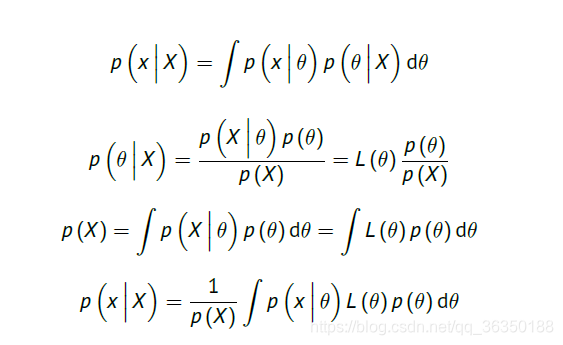
这里稍微对图里的参数进行解释:\
$p(\theta)$是估计参数的先验分布,它与给的数据集无关,而是我们的一种经验性判断。对于$p(\theta|X)$,是一个后验分布,所以一般取期望值,而且
举个例子:\
我们现在有高斯分布的数据集,其方差已知,我们要估计其均值,有如下关系:
对于先验我们的判断是:
根据采样均值和贝叶斯估计就能对均值进行估计:
最后这里补充个概念:
在贝叶斯统计中,如果后验分布与先验分布属于同类,则先验分布与后验分布被称为共轭分布,而先验分布被称为似然函数的共轭先验(Conjugate prior)。比如,高斯分布家族在高斯似然函数下与其自身共轭 (自共轭)。(来自 wiki百科)
比如上面那个均值的高斯先验,就是高斯模型的共轭,这里使用是因为:
- The product of two Gaussians is a Gaussian.\
- The marginal of a Gaussian is a Gaussian.
(附)参数估计的代码作业

简单来说就是给两组二维数据,模型是高斯分布,进行参数估计拟合,第一题是纯粹的统计,跳过,第二题的话。。。还是直接附代码了吧(代码正确性未知,因为作业还没有公布结果)1
2
3
4
5
6
7
8
9
10
11
12
13
14
15
16
17
18
19
20
21
22
23
24
25
26
27
28
29
30
31
32
33
34
35
36
37
38
39
40
41
42
43
44
45
46
47
48
49
50
51
52
53
54
55
56
57
58
59
60
61
62
63
64
65
66
67
68
69
70
71
72
73
74
75
76
77
78
79
80
81
82
83
84
85
86
87
88
89
90
91
92
93
94
95
96
97
98
99
100
101
102
103
104
105
106
107
108
109
110
111
112
113
114
115
116
117
118
119
120def load_data(path):
return np.loadtxt(path)
def prior_probability(arr_n, arr_sum):
h_n, w_n = arr_n.shape
h_sum, w_sum = arr_sum.shape
p_n = (h_n * w_n) / (h_sum * w_sum)
return p_n
def MLE_mean(arr):
return np.mean(arr, axis = 0)
def bias_unbias_var(arr):
num = arr.shape[0]
arr_mean = MLE_mean(arr)
dist = arr - arr_mean
sum_vor_cov = np.zeros((2, 2))
for i in range(num):
ls = []
ls.append(dist[i])
dist_arr = np.array(ls)
sum_vor_cov += np.dot(dist_arr.T, dist_arr)
return sum_vor_cov / num, sum_vor_cov / (num - 1)
def two_dim_Gauss(x, mu, cov):
#x = x.reshape(2, -1)
p_x = np.exp((-0.5*(x - mu) @ np.linalg.inv(cov) @ (x - mu).T)) \
/ np.sqrt((2 * np.pi) ** 2 * np.linalg.det(cov))
#print(p_x.shape)
return p_x
def posterior(mu1, mu2, cov1, cov2, p_c1, p_c2):
num = 300
x = np.linspace(-10, 10, num)
y = np.linspace(-10, 10, num)
X,Y = np.meshgrid(x, y)
C1 = []
C2 = []
for i in range(len(x)):
for j in range(len(y)):
z1 = two_dim_Gauss(np.array([[x[i], y[j]]]), mu1, cov1)
z2 = two_dim_Gauss(np.array([[x[i], y[j]]]), mu2, cov2)
temp1 = z1 * p_c1
temp2 = z2 * p_c2
if temp1 > temp2:
C1.append(np.array([[x[i], y[j]]]))
else:
C2.append(np.array([[x[i], y[j]]]))
C1 = np.array(C1).reshape(-1,2)
C2 = np.array(C2).reshape(-1,2)
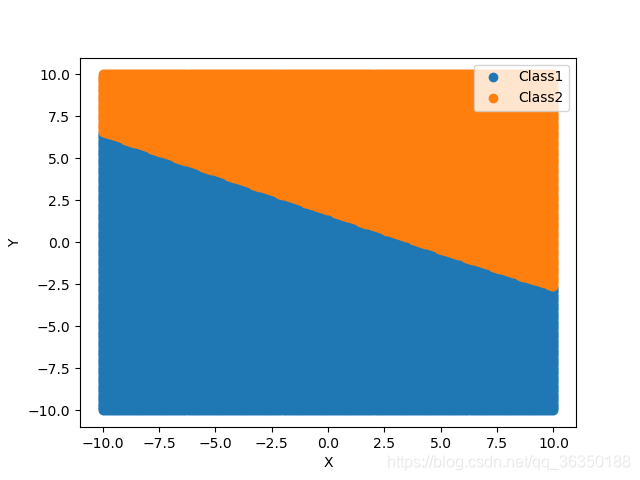
plt.scatter(C1[:,0], C1[:,1])
plt.scatter(C2[:,0], C2[:,1])
plt.xlabel("X")
plt.ylabel("Y")
plt.legend(['Class1', 'Class2'])
plt.show()
def plot_Gauss_point(arr, mu, cov):
plt.figure()
plt.scatter(arr[:,0], arr[:,1])
x_min, x_max = plt.gca().get_xlim()
y_min, y_max = plt.gca().get_ylim()
num = 50
x = np.linspace(x_min, x_max, num)
y = np.linspace(y_min, y_max, num)
X, Y = np.meshgrid(x, y)
Z = np.zeros_like(X)
for i in range(len(x)):
for j in range(len(y)):
z = two_dim_Gauss(np.array([[x[i], y[j]]]), mu, cov)
Z[j][i] = z
plt.contour(X,Y,Z)
plt.xlabel('X')
plt.ylabel('Y')
path1 = "./dataSets/densEst1.txt"
path2 = "./dataSets/densEst2.txt"
def main():
arr1 = load_data(path1)
arr2 = load_data(path2)
arr_sum = np.vstack((arr1, arr2))
p_C1 = prior_probability(arr1, arr_sum)
p_C2 = prior_probability(arr2, arr_sum)
mu2 = MLE_mean(arr2)
bias_s2, sigma2 = bias_unbias_var(arr2)
mu1 = MLE_mean(arr1)
bias_s1, sigma1 = bias_unbias_var(arr1)
print("The mean for C1 is: ", mu1)
print("The mean for C2 is: ", mu2)
print("The sigma with bias for C1 is", bias_s1)
print("The sigma without bias for C1 is", sigma1)
print("The sigma with bias for C2 is", bias_s2)
print("The sigma without bias for C2 is", sigma2)
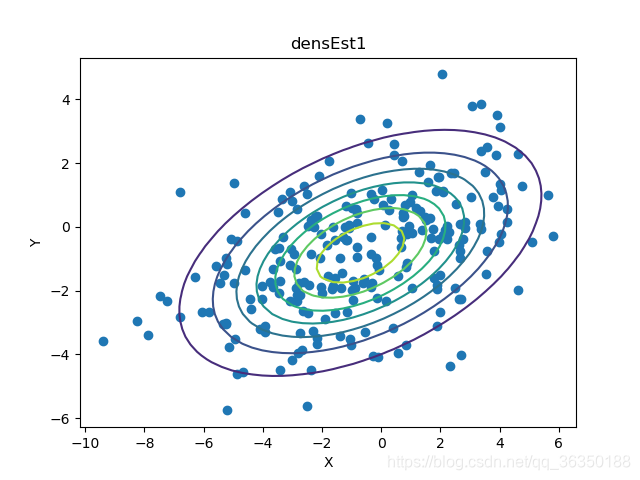
plot_Gauss_point(arr1, mu1, sigma1)
plt.title("densEst1")
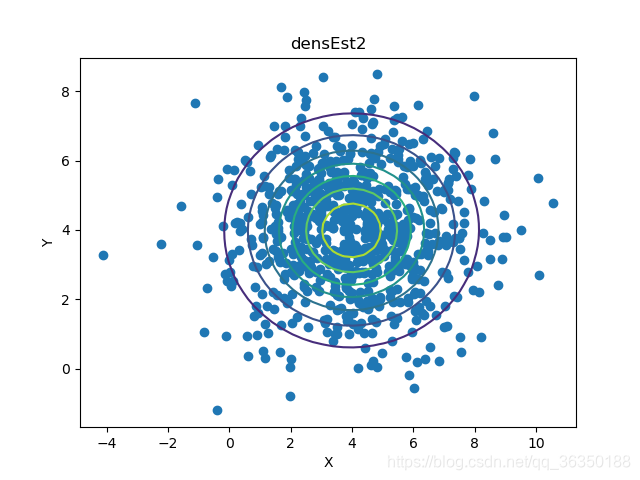
plot_Gauss_point(arr2, mu2, sigma2)
plt.title("densEst2")
plt.show()
posterior(mu1, mu2, sigma1, sigma2, p_C1, p_C2)
if __name__ == "__main__":
main()
结果如下: